7 Предобработка данных. Концепция tidy data
Мы приступаем к одной из ключевых тем курса — предобработка данных. И это тему мы будем осваивать c помощью семейства пакетов tidyverse.
7.1 tidyverse
Чтобы преуспеть в этом деле, нам надо поставить себе на машину пакет с пакетами:
Какие пакеты внутри пакета?
ggplot2— для визуализацииtibble— для работы с тибблами (как датафрейм, только лучше)tidyr— для формата tidy datareadr— для чтения файловpurrr— для функционального программированияdplyr— для преобразования данныхstringr— для работы со строковыми переменными (с ним мы уже виделись)forcats— для работы с факторными переменными
Кроме этого джентельменского набора нам еще пригодится пакет readxl, и которого мы будем брать функции для чтения файлов .xls и .xlsx.
Подключаем пакеты к сессии:
## ── Attaching packages ─────────────────────────────────────── tidyverse 1.3.0 ──## ✓ ggplot2 3.3.2 ✓ purrr 0.3.4
## ✓ tibble 3.0.4 ✓ dplyr 1.0.2
## ✓ tidyr 1.1.2 ✓ stringr 1.4.0
## ✓ readr 1.4.0 ✓ forcats 0.5.0## ── Conflicts ────────────────────────────────────────── tidyverse_conflicts() ──
## x dplyr::filter() masks stats::filter()
## x dplyr::lag() masks stats::lag()Нам написали верси пакетов, которые мы подгрузили, а также обозначили ряд конфликтов, которые произошли — базовые функции перекрылись функциями из внешних пакетов1. Но это всё можно игнорировать.
7.2 Импорт данных
Мы уже говорили с вами, что для импорта данных есть generic-функция read.table() и ещё ряд функций, которые адаптированы под конкретные форматы данных (read.tsv(), read.csv()). Это всё хорошо и приятно, но мы будем пользоваться их сестами-близнецами из пакета readr:
read_csv("path/to/file") # для чтения классических csv-файлов
read_csv2("path/to/file") # для чтения csv-файлов с разделителем точка в запятой
read_delim("path/to/file", delim = "...") # для чтения файлов с любым разделителемЭти функции, как и базовые, умеют открывать файлы
- только по имени (если файл лежит в рабочей директории)
- по относительному пути (если файл лежит в подпапке рабочей директории)
- по абсолютному пути (когда мы прописываем путь к файлу от корня)
- напрямую из интернета (при наличии интернет-соединения)
Каков профит? Во-первых, они работают быстрее, но на наших объемах данных мы этого, скорее всего, не заметим. А во-вторых, они считывают данные не в датафрейм, а в tibble, что очень радостно и приятно, если осознать, что tibble есть такое.
7.3 tibble
Мы уже умеет работать с датафреймами, однако мы пока не сталкивались с некоторыми их особенностями. В частности, если мы попытаемся создать новую переменную на основе имеющейся прям при создании датафрейма, случится ошибка. Пусть у нас есть колонка с возрастом респондентов, и мы хотим сделать новый переменную, которая будет задавать контрастные группы:
data.frame(age = sample(18:60, size = 30),
group = ifelse(age > 40, 'old',
ifelse(age < 25, 'young', 'middle')))## Error in ifelse(age > 40, "old", ifelse(age < 25, "young", "middle")): object 'age' not foundR говорит нам, что переменная age ещё не создана, что в целом достаточно справедливо. Мы можем эту напасть побороть, сначала создав переменную age, а затем сделать новую переменную group через присваивание… но чёт лень…
Лучше воспользуемся мощностями tibble:
tibble(age = sample(18:60, size = 30),
group = ifelse(age > 40, 'old',
ifelse(age < 25, 'young', 'middle')))## # A tibble: 30 x 2
## age group
## <int> <chr>
## 1 31 middle
## 2 40 middle
## 3 56 old
## 4 27 middle
## 5 52 old
## 6 55 old
## 7 29 middle
## 8 53 old
## 9 48 old
## 10 30 middle
## # … with 20 more rowsРоскошно!
Может показаться, что это достаточно незначительно преимущество. В целом, да — tibble и data.frame практически одно и то же. Однако, во-первых, стоит помнить, что функции из tidyverse всегда возвращают tibble, а во-вторых, когда мы познаем всю мощь тёмной стороны функции mutate(), мы осознаем полезность фичи исползования переменных «по ходу событий».
7.4 Грузим много данных
На самом деле, не так уж и много. Просто в этот раз они располагаются в разных файлах. Нам понадобится вот этот архив.
Его надо (1) разархивировать, и (2) положить всю получившуюся папку в нашу папку data в директории курса.
Кстати, чтобы отобразить список файлов, которые лежат в некоторой папке есть команда dir() (по умолчанию отображает содержимое рабочей директории):
## [1] "01.xlsx" "02.xlsx" "03.xlsx"
## [4] "04.xlsx" "05.xlsx" "06.xlsx"
## [7] "07.xlsx" "08.xlsx" "09.xlsx"
## [10] "10.xlsx" "11.xlsx" "12.xlsx"
## [13] "13.xlsx" "14.xlsx" "15.xlsx"
## [16] "16.xlsx" "17.xlsx" "18.xlsx"
## [19] "19.xlsx" "20.xlsx" "targetpositions.xlsx"Функция возвращает строковый вектор, поэтому чтобы узнать количество файлов в директории, можно сделать следующее:
## [1] 21У нас есть 21 файл. Это данные поведенческого эксперимента, в котором пользователи платформ iOS и Android искали иконки share обеих платформ среди других иконок. Судя по тому, как названы файлы, у нас есть 20 Excel-ек с данными и файл targetpositions.xlsx, в котором содержатся координаты целевых стимулов.
Загрузим один файл, и посмотрим, что получится. Нам будет нужен второй лист, так как данные основной серии записаны на нём:
## tibble [459 × 91] (S3: tbl_df/tbl/data.frame)
## $ stim1 : chr [1:459] "tray1.png" "08.png" NA "22.png" ...
## $ stim2 : chr [1:459] "01.png" "tray1.png" NA "23.png" ...
## $ stim3 : chr [1:459] "02.png" "09.png" "tray1.png" "24.png" ...
## $ stim4 : chr [1:459] "03.png" NA "15.png" "tray1.png" ...
## $ stim5 : chr [1:459] "04.png" "10.png" "16.png" NA ...
## $ stim6 : chr [1:459] "05.png" "11.png" "17.png" NA ...
## $ stim7 : chr [1:459] "06.png" "12.png" "18.png" "25.png" ...
## $ stim8 : chr [1:459] "07.png" "13.png" "19.png" "26.png" ...
## $ stim9 : chr [1:459] NA NA "20.png" NA ...
## $ stim10 : chr [1:459] NA "14.png" "21.png" NA ...
## $ stim11 : chr [1:459] NA NA NA "27.png" ...
## $ stim12 : chr [1:459] NA NA NA "28.png" ...
## $ stim13 : chr [1:459] NA NA NA NA ...
## $ stim14 : chr [1:459] NA NA NA NA ...
## $ stim15 : chr [1:459] NA NA NA NA ...
## $ stim16 : chr [1:459] NA NA NA NA ...
## $ pos1 : chr [1:459] "[-238, 202]" "[-213, 226]" "[-234, 217]" "[-247, 218]" ...
## $ pos2 : chr [1:459] "[-88, 202]" "[-63, 226]" "[-84, 217]" "[-97, 218]" ...
## $ pos3 : chr [1:459] "[62, 202]" "[87, 226]" "[66, 217]" "[53, 218]" ...
## $ pos4 : chr [1:459] "[212, 202]" "[237, 226]" "[216, 217]" "[203, 218]" ...
## $ pos5 : chr [1:459] "[-238, 52]" "[-213, 76]" "[-234, 67]" "[-247, 68]" ...
## $ pos6 : chr [1:459] "[-88, 52]" "[-63, 76]" "[-84, 67]" "[-97, 68]" ...
## $ pos7 : chr [1:459] "[62, 52]" "[87, 76]" "[66, 67]" "[53, 68]" ...
## $ pos8 : chr [1:459] "[212, 52]" "[237, 76]" "[216, 67]" "[203, 68]" ...
## $ pos9 : chr [1:459] "[-238, -98]" "[-213, -74]" "[-234, -83]" "[-247, -82]" ...
## $ pos10 : chr [1:459] "[-88, -98]" "[-63, -74]" "[-84, -83]" "[-97, -82]" ...
## $ pos11 : chr [1:459] "[62, -98]" "[87, -74]" "[66, -83]" "[53, -82]" ...
## $ pos12 : chr [1:459] "[212, -98]" "[237, -74]" "[216, -83]" "[203, -82]" ...
## $ pos13 : chr [1:459] "[-238, -248]" "[-213, -224]" "[-234, -233]" "[-247, -232]" ...
## $ pos14 : chr [1:459] "[-88, -248]" "[-63, -224]" "[-84, -233]" "[-97, -232]" ...
## $ pos15 : chr [1:459] "[62, -248]" "[87, -224]" "[66, -233]" "[53, -232]" ...
## $ pos16 : chr [1:459] "[212, -248]" "[237, -224]" "[216, -233]" "[203, -232]" ...
## $ size1 : chr [1:459] "[100, 100]" "[100, 100]" "[100, 100]" "[100, 100]" ...
## $ size2 : chr [1:459] "[100, 100]" "[100, 100]" "[100, 100]" "[100, 100]" ...
## $ size3 : chr [1:459] "[100, 100]" "[100, 100]" "[100, 100]" "[100, 100]" ...
## $ size4 : chr [1:459] "[100, 100]" "[100, 100]" "[100, 100]" "[100, 100]" ...
## $ size5 : chr [1:459] "[100, 100]" "[100, 100]" "[100, 100]" "[100, 100]" ...
## $ size6 : chr [1:459] "[100, 100]" "[100, 100]" "[100, 100]" "[100, 100]" ...
## $ size7 : chr [1:459] "[100, 100]" "[100, 100]" "[100, 100]" "[100, 100]" ...
## $ size8 : chr [1:459] "[100, 100]" "[100, 100]" "[100, 100]" "[100, 100]" ...
## $ size9 : chr [1:459] "[100, 100]" "[100, 100]" "[100, 100]" "[100, 100]" ...
## $ size10 : chr [1:459] "[100, 100]" "[100, 100]" "[100, 100]" "[100, 100]" ...
## $ size11 : chr [1:459] "[100, 100]" "[100, 100]" "[100, 100]" "[100, 100]" ...
## $ size12 : chr [1:459] "[100, 100]" "[100, 100]" "[100, 100]" "[100, 100]" ...
## $ size13 : chr [1:459] "[100, 100]" "[100, 100]" "[100, 100]" "[100, 100]" ...
## $ size14 : chr [1:459] "[100, 100]" "[100, 100]" "[100, 100]" "[100, 100]" ...
## $ size15 : chr [1:459] "[100, 100]" "[100, 100]" "[100, 100]" "[100, 100]" ...
## $ size16 : chr [1:459] "[100, 100]" "[100, 100]" "[100, 100]" "[100, 100]" ...
## $ ok : chr [1:459] "ok.png" "ok.png" "ok.png" "ok.png" ...
## $ posok : chr [1:459] "[0, -375]" "[0, -375]" "[0, -375]" "[0, -375]" ...
## $ sizeok : chr [1:459] "[150, 100]" "[150, 100]" "[150, 100]" "[150, 100]" ...
## $ numtrial : num [1:459] 1 2 3 4 5 6 7 8 9 10 ...
## $ trialtype : chr [1:459] "tray" "tray" "tray" "tray" ...
## $ setsize : num [1:459] 8 8 8 8 8 8 8 8 8 8 ...
## $ trialtypenum : num [1:459] 1 1 1 1 1 1 1 1 1 1 ...
## $ ser : chr [1:459] "m" "m" "m" "m" ...
## $ sernum : num [1:459] 1 1 1 1 1 1 1 1 1 1 ...
## $ n : num [1:459] 1 1 1 1 1 1 1 1 1 1 ...
## $ mouse_main1.leftButton_mean : num [1:459] 1 1 1 1 1 1 1 1 1 1 ...
## $ mouse_main1.leftButton_raw : num [1:459] 1 1 1 1 1 1 1 1 1 1 ...
## $ mouse_main1.leftButton_std : num [1:459] 0 0 0 0 0 0 0 0 0 0 ...
## $ mouse_main1.midButton_mean : num [1:459] 0 0 0 0 0 0 0 0 0 0 ...
## $ mouse_main1.midButton_raw : num [1:459] 0 0 0 0 0 0 0 0 0 0 ...
## $ mouse_main1.midButton_std : num [1:459] 0 0 0 0 0 0 0 0 0 0 ...
## $ mouse_main1.rightButton_mean: num [1:459] 0 0 0 0 0 0 0 0 0 0 ...
## $ mouse_main1.rightButton_raw : num [1:459] 0 0 0 0 0 0 0 0 0 0 ...
## $ mouse_main1.rightButton_std : num [1:459] 0 0 0 0 0 0 0 0 0 0 ...
## $ mouse_main1.time_mean : num [1:459] 1.67 1.13 2.6 2.61 1.59 ...
## $ mouse_main1.time_raw : num [1:459] 1.67 1.13 2.6 2.61 1.59 ...
## $ mouse_main1.time_std : num [1:459] 0 0 0 0 0 0 0 0 0 0 ...
## $ mouse_main1.x_mean : num [1:459] -227 -69 60 199 -241 -51 99 213 -201 -70 ...
## $ mouse_main1.x_raw : num [1:459] -227 -69 60 199 -241 -51 99 213 -201 -70 ...
## $ mouse_main1.y_mean : num [1:459] 202 231 195 213 43 59 62 46 -123 -82 ...
## $ mouse_main1.y_raw : num [1:459] 202 231 195 213 43 59 62 46 -123 -82 ...
## $ mouse_main2.leftButton_mean : num [1:459] 1 1 1 1 1 1 1 1 1 1 ...
## $ mouse_main2.leftButton_raw : num [1:459] 1 1 1 1 1 1 1 1 1 1 ...
## $ mouse_main2.leftButton_std : num [1:459] 0 0 0 0 0 0 0 0 0 0 ...
## $ mouse_main2.midButton_mean : num [1:459] 0 0 0 0 0 0 0 0 0 0 ...
## $ mouse_main2.midButton_raw : num [1:459] 0 0 0 0 0 0 0 0 0 0 ...
## $ mouse_main2.midButton_std : num [1:459] 0 0 0 0 0 0 0 0 0 0 ...
## $ mouse_main2.rightButton_mean: num [1:459] 0 0 0 0 0 0 0 0 0 0 ...
## $ mouse_main2.rightButton_raw : num [1:459] 0 0 0 0 0 0 0 0 0 0 ...
## $ mouse_main2.rightButton_std : num [1:459] 0 0 0 0 0 0 0 0 0 0 ...
## $ mouse_main2.time_mean : num [1:459] 1.28 1.061 0.963 0.863 0.931 ...
## $ mouse_main2.time_raw : num [1:459] 1.28 1.061 0.963 0.863 0.931 ...
## $ mouse_main2.time_std : num [1:459] 0 0 0 0 0 0 0 0 0 0 ...
## $ mouse_main2.x_mean : num [1:459] 14 -44 17 -26 -25 10 -29 -27 -25 -19 ...
## $ mouse_main2.x_raw : num [1:459] 14 -44 17 -26 -25 10 -29 -27 -25 -19 ...
## $ mouse_main2.y_mean : num [1:459] -351 -392 -361 -356 -397 -383 -372 -353 -385 -394 ...
## $ mouse_main2.y_raw : num [1:459] -351 -392 -361 -356 -397 -383 -372 -353 -385 -394 ...
## $ order : num [1:459] 353 441 247 364 246 45 411 257 273 252 ...Данные выглядят адекватно, но — куча переменных. Нам явно нужны не все.
7.5 Сабсет данных
Кто знаком с аутпутами PsychoPy, тот опознает смысл названий переменных. Кто не знаком с аутпутами PsychoPy — не страшно. Нам нужны следующие переменные:
- независимые переменные:
trialtype— тип пробы (tray,dots,both)setsize— количество стимулов в пробе (8,12,16)
- зависимые переменные (или те переменные, на основе которых мы их посчитаем):
mouse_main1.time_raw— время первого клика (мс)mouse_main1.x_raw— координатаxпервого кликаmouse_main1.y_raw— координатаyпервого кликаmouse_main2.time_raw— время второго клика (мс)mouse_main2.x_raw— координатаxвторого кликаmouse_main2.y_raw— координатаyвторого клика
numtrial— номер пробы в исходном файле с координатами (пригодится нам позднее)
Давайте их вытаскивать.
7.5.1 %>%
Для того, чтобы это делать элегантно и красиво, нам надо познакомиться с ключевой особенностью синтаксиса tidyverse. На секунду отвлечемся от данных и представим, что нам надо посчитать вот такое дикое выражение:
## [1] 0.3846181Запись верная, но читать это совершенно невозможно. Чтобы решить этот вопрос, нам на помощь приходит pipe — специальный оператор, чем-то напоминающий вилосипедик %>%.
Работает он очень просто: он передаёт то, что от него стоит слева, в функцию, которая стоит от него справа, в качестве первого аргумента. То есть эти две команды
## [1] 7## [1] 7содержательно абсолютно идентичны. Только записываются немного по-разному.
Если переписать выражение выше с помощью pipes, то получится очень понятный «конвейер»:
## [1] 0.3846181Теперь отчетливо видна последовательность функций, которые мы совершали, и более того, если что-то можно не так, то можно запросто откопать баг, выполнив только часть кода до определенного пайпа. Во второй строчке нам встретилась особенность — если нам нужно передать объект слева в функцию справа, но не первым аргументом, надо указать его позицию с помощью точки2.
Теперь мы готовы идти в бой!
7.5.2 select()
Фнукция select() выбирает опредлённые колонки из нашего датасета. Работает очень просто:
raw01 %>% select(trialtype,
setsize,
numtrial,
mouse_main1.time_raw,
mouse_main1.x_raw,
mouse_main1.y_raw,
mouse_main2.time_raw,
mouse_main2.x_raw,
mouse_main2.y_raw)## # A tibble: 459 x 9
## trialtype setsize numtrial mouse_main1.tim… mouse_main1.x_r… mouse_main1.y_r…
## <chr> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 tray 8 1 1.67 -227 202
## 2 tray 8 2 1.13 -69 231
## 3 tray 8 3 2.60 60 195
## 4 tray 8 4 2.61 199 213
## 5 tray 8 5 1.59 -241 43
## 6 tray 8 6 1.14 -51 59
## 7 tray 8 7 2.14 99 62
## 8 tray 8 8 1.63 213 46
## 9 tray 8 9 2.56 -201 -123
## 10 tray 8 10 2.02 -70 -82
## # … with 449 more rows, and 3 more variables: mouse_main2.time_raw <dbl>,
## # mouse_main2.x_raw <dbl>, mouse_main2.y_raw <dbl>Кайф! Только пока мы наш новый тиббл никуда не записали. Но, подождём — мы ещё не закончили.
7.5.3 rename()
Мы будем теперь практически всегда обращаться к колонкам по имени, поэтому полезно будет их сразу переименовать во что-то более юзабельное. Функци rename() занимается именно этим. В качестве её аргументов надо перечислить новые имена колонок и к каким колонкам они относятся:
raw01 %>% select(trialtype,
setsize,
numtrial,
mouse_main1.time_raw,
mouse_main1.x_raw,
mouse_main1.y_raw,
mouse_main2.time_raw,
mouse_main2.x_raw,
mouse_main2.y_raw) %>%
rename(time1 = mouse_main1.time_raw,
click1x = mouse_main1.x_raw,
click1y = mouse_main1.y_raw,
time2 = mouse_main2.time_raw,
click2x = mouse_main2.x_raw,
click2y = mouse_main2.y_raw)## # A tibble: 459 x 9
## trialtype setsize numtrial time1 click1x click1y time2 click2x click2y
## <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 tray 8 1 1.67 -227 202 1.28 14 -351
## 2 tray 8 2 1.13 -69 231 1.06 -44 -392
## 3 tray 8 3 2.60 60 195 0.963 17 -361
## 4 tray 8 4 2.61 199 213 0.863 -26 -356
## 5 tray 8 5 1.59 -241 43 0.931 -25 -397
## 6 tray 8 6 1.14 -51 59 0.896 10 -383
## 7 tray 8 7 2.14 99 62 0.879 -29 -372
## 8 tray 8 8 1.63 213 46 0.947 -27 -353
## 9 tray 8 9 2.56 -201 -123 1.65 -25 -385
## 10 tray 8 10 2.02 -70 -82 0.970 -19 -394
## # … with 449 more rowsКрасuво! Заметьте, что мы продолжаем наш «конвейер».
7.5.4 slice()
Посмотрим, если строки с пропущенными значениями. Прям тут, не отходя от конвейера:
raw01 %>% select(trialtype,
setsize,
numtrial,
mouse_main1.time_raw,
mouse_main1.x_raw,
mouse_main1.y_raw,
mouse_main2.time_raw,
mouse_main2.x_raw,
mouse_main2.y_raw) %>%
rename(time1 = mouse_main1.time_raw,
click1x = mouse_main1.x_raw,
click1y = mouse_main1.y_raw,
time2 = mouse_main2.time_raw,
click2x = mouse_main2.x_raw,
click2y = mouse_main2.y_raw) %>%
sapply(is.na) %>% apply(2, sum)## trialtype setsize numtrial time1 click1x click1y time2 click2x
## 9 9 9 9 9 9 9 9
## click2y
## 9В каждой переменной есть по девять пропусков. Чтобы понять, что это за пропуски, надо немного знать о том, как PsychoPy записывает данные. После того, как он написал в Excel все экспериментальные данные, он в самом низу дописывает соцдем, который испытуемые заполняют в самом начале эксперимента.
Вот эта штука:
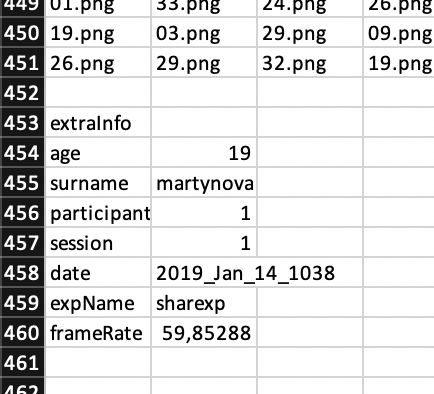
Как раз девять строчек — они нам и делают пропущенные значения. Так как они идут после всех наших данные, то мы можем просто «отрезать» этот кусок таблицы:
raw01 %>% select(trialtype,
setsize,
numtrial,
mouse_main1.time_raw,
mouse_main1.x_raw,
mouse_main1.y_raw,
mouse_main2.time_raw,
mouse_main2.x_raw,
mouse_main2.y_raw) %>%
rename(time1 = mouse_main1.time_raw,
click1x = mouse_main1.x_raw,
click1y = mouse_main1.y_raw,
time2 = mouse_main2.time_raw,
click2x = mouse_main2.x_raw,
click2y = mouse_main2.y_raw) %>%
slice(1:450) # мы знаем, что у нас было 450 экспериментальных проб## # A tibble: 450 x 9
## trialtype setsize numtrial time1 click1x click1y time2 click2x click2y
## <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 tray 8 1 1.67 -227 202 1.28 14 -351
## 2 tray 8 2 1.13 -69 231 1.06 -44 -392
## 3 tray 8 3 2.60 60 195 0.963 17 -361
## 4 tray 8 4 2.61 199 213 0.863 -26 -356
## 5 tray 8 5 1.59 -241 43 0.931 -25 -397
## 6 tray 8 6 1.14 -51 59 0.896 10 -383
## 7 tray 8 7 2.14 99 62 0.879 -29 -372
## 8 tray 8 8 1.63 213 46 0.947 -27 -353
## 9 tray 8 9 2.56 -201 -123 1.65 -25 -385
## 10 tray 8 10 2.02 -70 -82 0.970 -19 -394
## # … with 440 more rowsМожно перепроверить, не осталось ли у нас пропущенных значений, с помощью той же строчки, что и в предыдущем чанке.
7.5.5 filter()
Для анализа нам будут нужны не все пробы, а только из экспериметальных условий tray и dots. А как мы помним из описания переменные, экспериметальных условий было три — tray, dots и both. Надо отфильтровать это третье условие. Изи:
raw01 %>% select(trialtype,
setsize,
numtrial,
mouse_main1.time_raw,
mouse_main1.x_raw,
mouse_main1.y_raw,
mouse_main2.time_raw,
mouse_main2.x_raw,
mouse_main2.y_raw) %>%
rename(time1 = mouse_main1.time_raw,
click1x = mouse_main1.x_raw,
click1y = mouse_main1.y_raw,
time2 = mouse_main2.time_raw,
click2x = mouse_main2.x_raw,
click2y = mouse_main2.y_raw) %>%
slice(1:450) %>%
filter(trialtype != 'both')## # A tibble: 300 x 9
## trialtype setsize numtrial time1 click1x click1y time2 click2x click2y
## <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 tray 8 1 1.67 -227 202 1.28 14 -351
## 2 tray 8 2 1.13 -69 231 1.06 -44 -392
## 3 tray 8 3 2.60 60 195 0.963 17 -361
## 4 tray 8 4 2.61 199 213 0.863 -26 -356
## 5 tray 8 5 1.59 -241 43 0.931 -25 -397
## 6 tray 8 6 1.14 -51 59 0.896 10 -383
## 7 tray 8 7 2.14 99 62 0.879 -29 -372
## 8 tray 8 8 1.63 213 46 0.947 -27 -353
## 9 tray 8 9 2.56 -201 -123 1.65 -25 -385
## 10 tray 8 10 2.02 -70 -82 0.970 -19 -394
## # … with 290 more rowsВот теперь хорошо и красиво! Можно сохранить в новый объект!
raw01 %>% select(trialtype,
setsize,
numtrial,
mouse_main1.time_raw,
mouse_main1.x_raw,
mouse_main1.y_raw,
mouse_main2.time_raw,
mouse_main2.x_raw,
mouse_main2.y_raw) %>%
rename(time1 = mouse_main1.time_raw,
click1x = mouse_main1.x_raw,
click1y = mouse_main1.y_raw,
time2 = mouse_main2.time_raw,
click2x = mouse_main2.x_raw,
click2y = mouse_main2.y_raw) %>%
slice(1:450) %>%
filter(trialtype != 'both') -> d01
str(d01)## tibble [300 × 9] (S3: tbl_df/tbl/data.frame)
## $ trialtype: chr [1:300] "tray" "tray" "tray" "tray" ...
## $ setsize : num [1:300] 8 8 8 8 8 8 8 8 8 8 ...
## $ numtrial : num [1:300] 1 2 3 4 5 6 7 8 9 10 ...
## $ time1 : num [1:300] 1.67 1.13 2.6 2.61 1.59 ...
## $ click1x : num [1:300] -227 -69 60 199 -241 -51 99 213 -201 -70 ...
## $ click1y : num [1:300] 202 231 195 213 43 59 62 46 -123 -82 ...
## $ time2 : num [1:300] 1.28 1.061 0.963 0.863 0.931 ...
## $ click2x : num [1:300] 14 -44 17 -26 -25 10 -29 -27 -25 -19 ...
## $ click2y : num [1:300] -351 -392 -361 -356 -397 -383 -372 -353 -385 -394 ...Да, assignment слева направо тоже работает, и в данном случае это хорошо согласуется с логикой пайпов.
Всё хорошо, но в данном датасете нам не хватает двух вещей — id нашего испытуемого. Зачем? Во-первых, он может быть нужен для некоторых видов анализа, а кроме того, он нам пригодится чуть позже, когда мы будем возиться ещё с одной историей.
7.6 mutate()
Функция mutate() позволяет делать дикую кучу всего, но пока познакомимся с логикой её работы.
## tibble [300 × 10] (S3: tbl_df/tbl/data.frame)
## $ trialtype: chr [1:300] "tray" "tray" "tray" "tray" ...
## $ setsize : num [1:300] 8 8 8 8 8 8 8 8 8 8 ...
## $ numtrial : num [1:300] 1 2 3 4 5 6 7 8 9 10 ...
## $ time1 : num [1:300] 1.67 1.13 2.6 2.61 1.59 ...
## $ click1x : num [1:300] -227 -69 60 199 -241 -51 99 213 -201 -70 ...
## $ click1y : num [1:300] 202 231 195 213 43 59 62 46 -123 -82 ...
## $ time2 : num [1:300] 1.28 1.061 0.963 0.863 0.931 ...
## $ click2x : num [1:300] 14 -44 17 -26 -25 10 -29 -27 -25 -19 ...
## $ click2y : num [1:300] -351 -392 -361 -356 -397 -383 -372 -353 -385 -394 ...
## $ id : num [1:300] 1 1 1 1 1 1 1 1 1 1 ...То есть она просто создаёт нам новую переменную. Круто же.
Не забудем перезаписать нас тиббл:
7.7 Догрузим остальные данные
Вернёмся немного назад и вспомним, что у нас не один файл данных, а двадцать. При этом — так как это автоматически записанные файлы — они одинаково устроены. То есть, чтобы их предобработать, нам надо будет двадцать раз повторить один и те же операции. Ну, не будем же мы двадцать раз копировать тот абзац кода, который мы написали!
Конечно, нет. Мы завернем его в функцию.
import_data <- function(x, id) {
library(tidyr)
library(readr)
readxl::read_excel(x, 2) %>%
select(
trialtype,
setsize,
numtrial,
mouse_main1.time_raw,
mouse_main1.x_raw,
mouse_main1.y_raw,
mouse_main2.time_raw,
mouse_main2.x_raw,
mouse_main2.y_raw
) %>%
rename(
time1 = mouse_main1.time_raw,
click1x = mouse_main1.x_raw,
click1y = mouse_main1.y_raw,
time2 = mouse_main2.time_raw,
click2x = mouse_main2.x_raw,
click2y = mouse_main2.y_raw
) %>%
slice(1:450) %>%
filter(trialtype != 'both') %>%
mutate(id = id) %>%
return()
}Роскошь! Теперь у нас есть функция, которая грузит данные и сразу их предобрабатывает! Ну, не чудо ли!
Вот только эта функция у нас работает на одном датасете, то есть чтобы загрузить все двадцать, нам всё равно придется написать 20 одинаковых строк кода. Нерадужное мероприятие.
Но мы сделаем по-другому.
7.7.1 Соединение датафреймов I
Наша конечная цель — получить единый датасет, в котором будут содержаться все наблюдения по всем испытуемым. Мы уже добились того, что наша функция возвращает нам ровный и красивый tidy tibble. Нам осталось лишь поставить их друг на друга, чтобы получить то, что нам нужно.
7.7.1.1 bind_...()
Слепить два тиббла можно либо по столбцам, либо по строкам. Этим занимаются две функции: bind_cols() и bind_rows() соответственно. Что происходит, когда количество строк / столбцов одинаковое — понятно: таблички просто слепляются. А вот если это не так:
## Warning: The `x` argument of `as_tibble.matrix()` must have unique column names if `.name_repair` is omitted as of tibble 2.0.0.
## Using compatibility `.name_repair`.
## This warning is displayed once every 8 hours.
## Call `lifecycle::last_warnings()` to see where this warning was generated.## # A tibble: 7 x 4
## V1 V2 V3 V4
## <int> <int> <int> <int>
## 1 1 4 7 10
## 2 2 5 8 11
## 3 3 6 9 12
## 4 1 5 9 NA
## 5 2 6 10 NA
## 6 3 7 11 NA
## 7 4 8 12 NA## Error: Can't recycle `..1` (size 3) to match `..2` (size 4).Вот так вот.
В нашем случае всё прозаично — нам просто нужно поставить 20 тибблов друг на друга. Нам — единственный раз! — понадобиться цикл.
share <- tibble() # заглушка для первой итерации
files <- paste0('data/data_sharexp/', dir('data/data_sharexp')[-21]) # вектор с названиями файлов
for (i in 1:20) {
import_data(files[i], i) %>% bind_rows(share, .) -> share
}Посмотрим, что получилось:
## tibble [6,000 × 10] (S3: tbl_df/tbl/data.frame)
## $ trialtype: chr [1:6000] "tray" "tray" "tray" "tray" ...
## $ setsize : num [1:6000] 8 8 8 8 8 8 8 8 8 8 ...
## $ numtrial : num [1:6000] 1 2 3 4 5 6 7 8 9 10 ...
## $ time1 : num [1:6000] 1.67 1.13 2.6 2.61 1.59 ...
## $ click1x : num [1:6000] -227 -69 60 199 -241 -51 99 213 -201 -70 ...
## $ click1y : num [1:6000] 202 231 195 213 43 59 62 46 -123 -82 ...
## $ time2 : num [1:6000] 1.28 1.061 0.963 0.863 0.931 ...
## $ click2x : num [1:6000] 14 -44 17 -26 -25 10 -29 -27 -25 -19 ...
## $ click2y : num [1:6000] -351 -392 -361 -356 -397 -383 -372 -353 -385 -394 ...
## $ id : int [1:6000] 1 1 1 1 1 1 1 1 1 1 ...Выглядит вроде верно.
## [1] 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20##
## 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18
## dots 150 150 150 150 150 150 150 150 150 150 150 150 150 150 150 150 150 150
## tray 150 150 150 150 150 150 150 150 150 150 150 150 150 150 150 150 150 150
##
## 19 20
## dots 150 150
## tray 150 150Вроде всё ровно. Чилл.
7.8 Сортировка
7.8.1 sort()
Иногда нам может потребоваться отсортировать наши данные. Как сортировать отдельные векторы, мы уже знаем:
## [1] 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1
## [19] 1 1 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2
## [37] 2 2 2 2 3 3 3 3 3 3 3 3 3 3 3 3 3 3
## [55] 3 3 3 3 3 3 4 4 4 4 4 4 4 4 4 4 4 4
## [73] 4 4 4 4 4 4 4 4 5 5 5 5 5 5 5 5 5 5
## [91] 5 5 5 5 5 5 5 5 5 5 6 6 6 6 6 6 6 6
## [109] 6 6 6 6 6 6 6 6 6 6 6 6 7 7 7 7 7 7
## [127] 7 7 7 7 7 7 7 7 7 7 7 7 7 7 8 8 8 8
## [145] 8 8 8 8 8 8 8 8 8 8 8 8 8 8 8 8 9 9
## [163] 9 9 9 9 9 9 9 9 9 9 9 9 9 9 9 9 9 9
## [181] 10 10 10 10 10 10 10 10 10 10 10 10 10 10 10 10 10 10
## [199] 10 10 11 11 11 11 11 11 11 11 11 11 11 11 11 11 11 11
## [217] 11 11 11 11 12 12 12 12 12 12 12 12 12 12 12 12 12 12
## [235] 12 12 12 12 12 12 13 13 13 13 13 13 13 13 13 13 13 13
## [253] 13 13 13 13 13 13 13 13 14 14 14 14 14 14 14 14 14 14
## [271] 14 14 14 14 14 14 14 14 14 14 15 15 15 15 15 15 15 15
## [289] 15 15 15 15 15 15 15 15 15 15 15 15 16 16 16 16 16 16
## [307] 16 16 16 16 16 16 16 16 16 16 16 16 16 16 17 17 17 17
## [325] 17 17 17 17 17 17 17 17 17 17 17 17 17 17 17 17 18 18
## [343] 18 18 18 18 18 18 18 18 18 18 18 18 18 18 18 18 18 18
## [361] 19 19 19 19 19 19 19 19 19 19 19 19 19 19 19 19 19 19
## [379] 19 19 20 20 20 20 20 20 20 20 20 20 20 20 20 20 20 20
## [397] 20 20 20 20 21 21 21 21 21 21 21 21 21 21 21 21 21 21
## [415] 21 21 21 21 21 21 22 22 22 22 22 22 22 22 22 22 22 22
## [433] 22 22 22 22 22 22 22 22 23 23 23 23 23 23 23 23 23 23
## [451] 23 23 23 23 23 23 23 23 23 23 24 24 24 24 24 24 24 24
## [469] 24 24 24 24 24 24 24 24 24 24 24 24 25 25 25 25 25 25
## [487] 25 25 25 25 25 25 25 25 25 25 25 25 25 25 26 26 26 26
## [505] 26 26 26 26 26 26 26 26 26 26 26 26 26 26 26 26 27 27
## [523] 27 27 27 27 27 27 27 27 27 27 27 27 27 27 27 27 27 27
## [541] 28 28 28 28 28 28 28 28 28 28 28 28 28 28 28 28 28 28
## [559] 28 28 29 29 29 29 29 29 29 29 29 29 29 29 29 29 29 29
## [577] 29 29 29 29 30 30 30 30 30 30 30 30 30 30 30 30 30 30
## [595] 30 30 30 30 30 30 31 31 31 31 31 31 31 31 31 31 31 31
## [613] 31 31 31 31 31 31 31 31 32 32 32 32 32 32 32 32 32 32
## [631] 32 32 32 32 32 32 32 32 32 32 33 33 33 33 33 33 33 33
## [649] 33 33 33 33 33 33 33 33 33 33 33 33 34 34 34 34 34 34
## [667] 34 34 34 34 34 34 34 34 34 34 34 34 34 34 35 35 35 35
## [685] 35 35 35 35 35 35 35 35 35 35 35 35 35 35 35 35 36 36
## [703] 36 36 36 36 36 36 36 36 36 36 36 36 36 36 36 36 36 36
## [721] 37 37 37 37 37 37 37 37 37 37 37 37 37 37 37 37 37 37
## [739] 37 37 38 38 38 38 38 38 38 38 38 38 38 38 38 38 38 38
## [757] 38 38 38 38 39 39 39 39 39 39 39 39 39 39 39 39 39 39
## [775] 39 39 39 39 39 39 40 40 40 40 40 40 40 40 40 40 40 40
## [793] 40 40 40 40 40 40 40 40 41 41 41 41 41 41 41 41 41 41
## [811] 41 41 41 41 41 41 41 41 41 41 42 42 42 42 42 42 42 42
## [829] 42 42 42 42 42 42 42 42 42 42 42 42 43 43 43 43 43 43
## [847] 43 43 43 43 43 43 43 43 43 43 43 43 43 43 44 44 44 44
## [865] 44 44 44 44 44 44 44 44 44 44 44 44 44 44 44 44 45 45
## [883] 45 45 45 45 45 45 45 45 45 45 45 45 45 45 45 45 45 45
## [901] 46 46 46 46 46 46 46 46 46 46 46 46 46 46 46 46 46 46
## [919] 46 46 47 47 47 47 47 47 47 47 47 47 47 47 47 47 47 47
## [937] 47 47 47 47 48 48 48 48 48 48 48 48 48 48 48 48 48 48
## [955] 48 48 48 48 48 48 49 49 49 49 49 49 49 49 49 49 49 49
## [973] 49 49 49 49 49 49 49 49 50 50 50 50 50 50 50 50 50 50
## [991] 50 50 50 50 50 50 50 50 50 50 51 51 51 51 51 51 51 51
## [1009] 51 51 51 51 51 51 51 51 51 51 51 51 52 52 52 52 52 52
## [1027] 52 52 52 52 52 52 52 52 52 52 52 52 52 52 53 53 53 53
## [1045] 53 53 53 53 53 53 53 53 53 53 53 53 53 53 53 53 54 54
## [1063] 54 54 54 54 54 54 54 54 54 54 54 54 54 54 54 54 54 54
## [1081] 55 55 55 55 55 55 55 55 55 55 55 55 55 55 55 55 55 55
## [1099] 55 55 56 56 56 56 56 56 56 56 56 56 56 56 56 56 56 56
## [1117] 56 56 56 56 57 57 57 57 57 57 57 57 57 57 57 57 57 57
## [1135] 57 57 57 57 57 57 58 58 58 58 58 58 58 58 58 58 58 58
## [1153] 58 58 58 58 58 58 58 58 59 59 59 59 59 59 59 59 59 59
## [1171] 59 59 59 59 59 59 59 59 59 59 60 60 60 60 60 60 60 60
## [1189] 60 60 60 60 60 60 60 60 60 60 60 60 61 61 61 61 61 61
## [1207] 61 61 61 61 61 61 61 61 61 61 61 61 61 61 62 62 62 62
## [1225] 62 62 62 62 62 62 62 62 62 62 62 62 62 62 62 62 63 63
## [1243] 63 63 63 63 63 63 63 63 63 63 63 63 63 63 63 63 63 63
## [1261] 64 64 64 64 64 64 64 64 64 64 64 64 64 64 64 64 64 64
## [1279] 64 64 65 65 65 65 65 65 65 65 65 65 65 65 65 65 65 65
## [1297] 65 65 65 65 66 66 66 66 66 66 66 66 66 66 66 66 66 66
## [1315] 66 66 66 66 66 66 67 67 67 67 67 67 67 67 67 67 67 67
## [1333] 67 67 67 67 67 67 67 67 68 68 68 68 68 68 68 68 68 68
## [1351] 68 68 68 68 68 68 68 68 68 68 69 69 69 69 69 69 69 69
## [1369] 69 69 69 69 69 69 69 69 69 69 69 69 70 70 70 70 70 70
## [1387] 70 70 70 70 70 70 70 70 70 70 70 70 70 70 71 71 71 71
## [1405] 71 71 71 71 71 71 71 71 71 71 71 71 71 71 71 71 72 72
## [1423] 72 72 72 72 72 72 72 72 72 72 72 72 72 72 72 72 72 72
## [1441] 73 73 73 73 73 73 73 73 73 73 73 73 73 73 73 73 73 73
## [1459] 73 73 74 74 74 74 74 74 74 74 74 74 74 74 74 74 74 74
## [1477] 74 74 74 74 75 75 75 75 75 75 75 75 75 75 75 75 75 75
## [1495] 75 75 75 75 75 75 76 76 76 76 76 76 76 76 76 76 76 76
## [1513] 76 76 76 76 76 76 76 76 77 77 77 77 77 77 77 77 77 77
## [1531] 77 77 77 77 77 77 77 77 77 77 78 78 78 78 78 78 78 78
## [1549] 78 78 78 78 78 78 78 78 78 78 78 78 79 79 79 79 79 79
## [1567] 79 79 79 79 79 79 79 79 79 79 79 79 79 79 80 80 80 80
## [1585] 80 80 80 80 80 80 80 80 80 80 80 80 80 80 80 80 81 81
## [1603] 81 81 81 81 81 81 81 81 81 81 81 81 81 81 81 81 81 81
## [1621] 82 82 82 82 82 82 82 82 82 82 82 82 82 82 82 82 82 82
## [1639] 82 82 83 83 83 83 83 83 83 83 83 83 83 83 83 83 83 83
## [1657] 83 83 83 83 84 84 84 84 84 84 84 84 84 84 84 84 84 84
## [1675] 84 84 84 84 84 84 85 85 85 85 85 85 85 85 85 85 85 85
## [1693] 85 85 85 85 85 85 85 85 86 86 86 86 86 86 86 86 86 86
## [1711] 86 86 86 86 86 86 86 86 86 86 87 87 87 87 87 87 87 87
## [1729] 87 87 87 87 87 87 87 87 87 87 87 87 88 88 88 88 88 88
## [1747] 88 88 88 88 88 88 88 88 88 88 88 88 88 88 89 89 89 89
## [1765] 89 89 89 89 89 89 89 89 89 89 89 89 89 89 89 89 90 90
## [1783] 90 90 90 90 90 90 90 90 90 90 90 90 90 90 90 90 90 90
## [1801] 91 91 91 91 91 91 91 91 91 91 91 91 91 91 91 91 91 91
## [1819] 91 91 92 92 92 92 92 92 92 92 92 92 92 92 92 92 92 92
## [1837] 92 92 92 92 93 93 93 93 93 93 93 93 93 93 93 93 93 93
## [1855] 93 93 93 93 93 93 94 94 94 94 94 94 94 94 94 94 94 94
## [1873] 94 94 94 94 94 94 94 94 95 95 95 95 95 95 95 95 95 95
## [1891] 95 95 95 95 95 95 95 95 95 95 96 96 96 96 96 96 96 96
## [1909] 96 96 96 96 96 96 96 96 96 96 96 96 97 97 97 97 97 97
## [1927] 97 97 97 97 97 97 97 97 97 97 97 97 97 97 98 98 98 98
## [1945] 98 98 98 98 98 98 98 98 98 98 98 98 98 98 98 98 99 99
## [1963] 99 99 99 99 99 99 99 99 99 99 99 99 99 99 99 99 99 99
## [1981] 100 100 100 100 100 100 100 100 100 100 100 100 100 100 100 100 100 100
## [1999] 100 100 151 151 151 151 151 151 151 151 151 151 151 151 151 151 151 151
## [2017] 151 151 151 151 152 152 152 152 152 152 152 152 152 152 152 152 152 152
## [2035] 152 152 152 152 152 152 153 153 153 153 153 153 153 153 153 153 153 153
## [2053] 153 153 153 153 153 153 153 153 154 154 154 154 154 154 154 154 154 154
## [2071] 154 154 154 154 154 154 154 154 154 154 155 155 155 155 155 155 155 155
## [2089] 155 155 155 155 155 155 155 155 155 155 155 155 156 156 156 156 156 156
## [2107] 156 156 156 156 156 156 156 156 156 156 156 156 156 156 157 157 157 157
## [2125] 157 157 157 157 157 157 157 157 157 157 157 157 157 157 157 157 158 158
## [2143] 158 158 158 158 158 158 158 158 158 158 158 158 158 158 158 158 158 158
## [2161] 159 159 159 159 159 159 159 159 159 159 159 159 159 159 159 159 159 159
## [2179] 159 159 160 160 160 160 160 160 160 160 160 160 160 160 160 160 160 160
## [2197] 160 160 160 160 161 161 161 161 161 161 161 161 161 161 161 161 161 161
## [2215] 161 161 161 161 161 161 162 162 162 162 162 162 162 162 162 162 162 162
## [2233] 162 162 162 162 162 162 162 162 163 163 163 163 163 163 163 163 163 163
## [2251] 163 163 163 163 163 163 163 163 163 163 164 164 164 164 164 164 164 164
## [2269] 164 164 164 164 164 164 164 164 164 164 164 164 165 165 165 165 165 165
## [2287] 165 165 165 165 165 165 165 165 165 165 165 165 165 165 166 166 166 166
## [2305] 166 166 166 166 166 166 166 166 166 166 166 166 166 166 166 166 167 167
## [2323] 167 167 167 167 167 167 167 167 167 167 167 167 167 167 167 167 167 167
## [2341] 168 168 168 168 168 168 168 168 168 168 168 168 168 168 168 168 168 168
## [2359] 168 168 169 169 169 169 169 169 169 169 169 169 169 169 169 169 169 169
## [2377] 169 169 169 169 170 170 170 170 170 170 170 170 170 170 170 170 170 170
## [2395] 170 170 170 170 170 170 171 171 171 171 171 171 171 171 171 171 171 171
## [2413] 171 171 171 171 171 171 171 171 172 172 172 172 172 172 172 172 172 172
## [2431] 172 172 172 172 172 172 172 172 172 172 173 173 173 173 173 173 173 173
## [2449] 173 173 173 173 173 173 173 173 173 173 173 173 174 174 174 174 174 174
## [2467] 174 174 174 174 174 174 174 174 174 174 174 174 174 174 175 175 175 175
## [2485] 175 175 175 175 175 175 175 175 175 175 175 175 175 175 175 175 176 176
## [2503] 176 176 176 176 176 176 176 176 176 176 176 176 176 176 176 176 176 176
## [2521] 177 177 177 177 177 177 177 177 177 177 177 177 177 177 177 177 177 177
## [2539] 177 177 178 178 178 178 178 178 178 178 178 178 178 178 178 178 178 178
## [2557] 178 178 178 178 179 179 179 179 179 179 179 179 179 179 179 179 179 179
## [2575] 179 179 179 179 179 179 180 180 180 180 180 180 180 180 180 180 180 180
## [2593] 180 180 180 180 180 180 180 180 181 181 181 181 181 181 181 181 181 181
## [2611] 181 181 181 181 181 181 181 181 181 181 182 182 182 182 182 182 182 182
## [2629] 182 182 182 182 182 182 182 182 182 182 182 182 183 183 183 183 183 183
## [2647] 183 183 183 183 183 183 183 183 183 183 183 183 183 183 184 184 184 184
## [2665] 184 184 184 184 184 184 184 184 184 184 184 184 184 184 184 184 185 185
## [2683] 185 185 185 185 185 185 185 185 185 185 185 185 185 185 185 185 185 185
## [2701] 186 186 186 186 186 186 186 186 186 186 186 186 186 186 186 186 186 186
## [2719] 186 186 187 187 187 187 187 187 187 187 187 187 187 187 187 187 187 187
## [2737] 187 187 187 187 188 188 188 188 188 188 188 188 188 188 188 188 188 188
## [2755] 188 188 188 188 188 188 189 189 189 189 189 189 189 189 189 189 189 189
## [2773] 189 189 189 189 189 189 189 189 190 190 190 190 190 190 190 190 190 190
## [2791] 190 190 190 190 190 190 190 190 190 190 191 191 191 191 191 191 191 191
## [2809] 191 191 191 191 191 191 191 191 191 191 191 191 192 192 192 192 192 192
## [2827] 192 192 192 192 192 192 192 192 192 192 192 192 192 192 193 193 193 193
## [2845] 193 193 193 193 193 193 193 193 193 193 193 193 193 193 193 193 194 194
## [2863] 194 194 194 194 194 194 194 194 194 194 194 194 194 194 194 194 194 194
## [2881] 195 195 195 195 195 195 195 195 195 195 195 195 195 195 195 195 195 195
## [2899] 195 195 196 196 196 196 196 196 196 196 196 196 196 196 196 196 196 196
## [2917] 196 196 196 196 197 197 197 197 197 197 197 197 197 197 197 197 197 197
## [2935] 197 197 197 197 197 197 198 198 198 198 198 198 198 198 198 198 198 198
## [2953] 198 198 198 198 198 198 198 198 199 199 199 199 199 199 199 199 199 199
## [2971] 199 199 199 199 199 199 199 199 199 199 200 200 200 200 200 200 200 200
## [2989] 200 200 200 200 200 200 200 200 200 200 200 200 201 201 201 201 201 201
## [3007] 201 201 201 201 201 201 201 201 201 201 201 201 201 201 202 202 202 202
## [3025] 202 202 202 202 202 202 202 202 202 202 202 202 202 202 202 202 203 203
## [3043] 203 203 203 203 203 203 203 203 203 203 203 203 203 203 203 203 203 203
## [3061] 204 204 204 204 204 204 204 204 204 204 204 204 204 204 204 204 204 204
## [3079] 204 204 205 205 205 205 205 205 205 205 205 205 205 205 205 205 205 205
## [3097] 205 205 205 205 206 206 206 206 206 206 206 206 206 206 206 206 206 206
## [3115] 206 206 206 206 206 206 207 207 207 207 207 207 207 207 207 207 207 207
## [3133] 207 207 207 207 207 207 207 207 208 208 208 208 208 208 208 208 208 208
## [3151] 208 208 208 208 208 208 208 208 208 208 209 209 209 209 209 209 209 209
## [3169] 209 209 209 209 209 209 209 209 209 209 209 209 210 210 210 210 210 210
## [3187] 210 210 210 210 210 210 210 210 210 210 210 210 210 210 211 211 211 211
## [3205] 211 211 211 211 211 211 211 211 211 211 211 211 211 211 211 211 212 212
## [3223] 212 212 212 212 212 212 212 212 212 212 212 212 212 212 212 212 212 212
## [3241] 213 213 213 213 213 213 213 213 213 213 213 213 213 213 213 213 213 213
## [3259] 213 213 214 214 214 214 214 214 214 214 214 214 214 214 214 214 214 214
## [3277] 214 214 214 214 215 215 215 215 215 215 215 215 215 215 215 215 215 215
## [3295] 215 215 215 215 215 215 216 216 216 216 216 216 216 216 216 216 216 216
## [3313] 216 216 216 216 216 216 216 216 217 217 217 217 217 217 217 217 217 217
## [3331] 217 217 217 217 217 217 217 217 217 217 218 218 218 218 218 218 218 218
## [3349] 218 218 218 218 218 218 218 218 218 218 218 218 219 219 219 219 219 219
## [3367] 219 219 219 219 219 219 219 219 219 219 219 219 219 219 220 220 220 220
## [3385] 220 220 220 220 220 220 220 220 220 220 220 220 220 220 220 220 221 221
## [3403] 221 221 221 221 221 221 221 221 221 221 221 221 221 221 221 221 221 221
## [3421] 222 222 222 222 222 222 222 222 222 222 222 222 222 222 222 222 222 222
## [3439] 222 222 223 223 223 223 223 223 223 223 223 223 223 223 223 223 223 223
## [3457] 223 223 223 223 224 224 224 224 224 224 224 224 224 224 224 224 224 224
## [3475] 224 224 224 224 224 224 225 225 225 225 225 225 225 225 225 225 225 225
## [3493] 225 225 225 225 225 225 225 225 226 226 226 226 226 226 226 226 226 226
## [3511] 226 226 226 226 226 226 226 226 226 226 227 227 227 227 227 227 227 227
## [3529] 227 227 227 227 227 227 227 227 227 227 227 227 228 228 228 228 228 228
## [3547] 228 228 228 228 228 228 228 228 228 228 228 228 228 228 229 229 229 229
## [3565] 229 229 229 229 229 229 229 229 229 229 229 229 229 229 229 229 230 230
## [3583] 230 230 230 230 230 230 230 230 230 230 230 230 230 230 230 230 230 230
## [3601] 231 231 231 231 231 231 231 231 231 231 231 231 231 231 231 231 231 231
## [3619] 231 231 232 232 232 232 232 232 232 232 232 232 232 232 232 232 232 232
## [3637] 232 232 232 232 233 233 233 233 233 233 233 233 233 233 233 233 233 233
## [3655] 233 233 233 233 233 233 234 234 234 234 234 234 234 234 234 234 234 234
## [3673] 234 234 234 234 234 234 234 234 235 235 235 235 235 235 235 235 235 235
## [3691] 235 235 235 235 235 235 235 235 235 235 236 236 236 236 236 236 236 236
## [3709] 236 236 236 236 236 236 236 236 236 236 236 236 237 237 237 237 237 237
## [3727] 237 237 237 237 237 237 237 237 237 237 237 237 237 237 238 238 238 238
## [3745] 238 238 238 238 238 238 238 238 238 238 238 238 238 238 238 238 239 239
## [3763] 239 239 239 239 239 239 239 239 239 239 239 239 239 239 239 239 239 239
## [3781] 240 240 240 240 240 240 240 240 240 240 240 240 240 240 240 240 240 240
## [3799] 240 240 241 241 241 241 241 241 241 241 241 241 241 241 241 241 241 241
## [3817] 241 241 241 241 242 242 242 242 242 242 242 242 242 242 242 242 242 242
## [3835] 242 242 242 242 242 242 243 243 243 243 243 243 243 243 243 243 243 243
## [3853] 243 243 243 243 243 243 243 243 244 244 244 244 244 244 244 244 244 244
## [3871] 244 244 244 244 244 244 244 244 244 244 245 245 245 245 245 245 245 245
## [3889] 245 245 245 245 245 245 245 245 245 245 245 245 246 246 246 246 246 246
## [3907] 246 246 246 246 246 246 246 246 246 246 246 246 246 246 247 247 247 247
## [3925] 247 247 247 247 247 247 247 247 247 247 247 247 247 247 247 247 248 248
## [3943] 248 248 248 248 248 248 248 248 248 248 248 248 248 248 248 248 248 248
## [3961] 249 249 249 249 249 249 249 249 249 249 249 249 249 249 249 249 249 249
## [3979] 249 249 250 250 250 250 250 250 250 250 250 250 250 250 250 250 250 250
## [3997] 250 250 250 250 301 301 301 301 301 301 301 301 301 301 301 301 301 301
## [4015] 301 301 301 301 301 301 302 302 302 302 302 302 302 302 302 302 302 302
## [4033] 302 302 302 302 302 302 302 302 303 303 303 303 303 303 303 303 303 303
## [4051] 303 303 303 303 303 303 303 303 303 303 304 304 304 304 304 304 304 304
## [4069] 304 304 304 304 304 304 304 304 304 304 304 304 305 305 305 305 305 305
## [4087] 305 305 305 305 305 305 305 305 305 305 305 305 305 305 306 306 306 306
## [4105] 306 306 306 306 306 306 306 306 306 306 306 306 306 306 306 306 307 307
## [4123] 307 307 307 307 307 307 307 307 307 307 307 307 307 307 307 307 307 307
## [4141] 308 308 308 308 308 308 308 308 308 308 308 308 308 308 308 308 308 308
## [4159] 308 308 309 309 309 309 309 309 309 309 309 309 309 309 309 309 309 309
## [4177] 309 309 309 309 310 310 310 310 310 310 310 310 310 310 310 310 310 310
## [4195] 310 310 310 310 310 310 311 311 311 311 311 311 311 311 311 311 311 311
## [4213] 311 311 311 311 311 311 311 311 312 312 312 312 312 312 312 312 312 312
## [4231] 312 312 312 312 312 312 312 312 312 312 313 313 313 313 313 313 313 313
## [4249] 313 313 313 313 313 313 313 313 313 313 313 313 314 314 314 314 314 314
## [4267] 314 314 314 314 314 314 314 314 314 314 314 314 314 314 315 315 315 315
## [4285] 315 315 315 315 315 315 315 315 315 315 315 315 315 315 315 315 316 316
## [4303] 316 316 316 316 316 316 316 316 316 316 316 316 316 316 316 316 316 316
## [4321] 317 317 317 317 317 317 317 317 317 317 317 317 317 317 317 317 317 317
## [4339] 317 317 318 318 318 318 318 318 318 318 318 318 318 318 318 318 318 318
## [4357] 318 318 318 318 319 319 319 319 319 319 319 319 319 319 319 319 319 319
## [4375] 319 319 319 319 319 319 320 320 320 320 320 320 320 320 320 320 320 320
## [4393] 320 320 320 320 320 320 320 320 321 321 321 321 321 321 321 321 321 321
## [4411] 321 321 321 321 321 321 321 321 321 321 322 322 322 322 322 322 322 322
## [4429] 322 322 322 322 322 322 322 322 322 322 322 322 323 323 323 323 323 323
## [4447] 323 323 323 323 323 323 323 323 323 323 323 323 323 323 324 324 324 324
## [4465] 324 324 324 324 324 324 324 324 324 324 324 324 324 324 324 324 325 325
## [4483] 325 325 325 325 325 325 325 325 325 325 325 325 325 325 325 325 325 325
## [4501] 326 326 326 326 326 326 326 326 326 326 326 326 326 326 326 326 326 326
## [4519] 326 326 327 327 327 327 327 327 327 327 327 327 327 327 327 327 327 327
## [4537] 327 327 327 327 328 328 328 328 328 328 328 328 328 328 328 328 328 328
## [4555] 328 328 328 328 328 328 329 329 329 329 329 329 329 329 329 329 329 329
## [4573] 329 329 329 329 329 329 329 329 330 330 330 330 330 330 330 330 330 330
## [4591] 330 330 330 330 330 330 330 330 330 330 331 331 331 331 331 331 331 331
## [4609] 331 331 331 331 331 331 331 331 331 331 331 331 332 332 332 332 332 332
## [4627] 332 332 332 332 332 332 332 332 332 332 332 332 332 332 333 333 333 333
## [4645] 333 333 333 333 333 333 333 333 333 333 333 333 333 333 333 333 334 334
## [4663] 334 334 334 334 334 334 334 334 334 334 334 334 334 334 334 334 334 334
## [4681] 335 335 335 335 335 335 335 335 335 335 335 335 335 335 335 335 335 335
## [4699] 335 335 336 336 336 336 336 336 336 336 336 336 336 336 336 336 336 336
## [4717] 336 336 336 336 337 337 337 337 337 337 337 337 337 337 337 337 337 337
## [4735] 337 337 337 337 337 337 338 338 338 338 338 338 338 338 338 338 338 338
## [4753] 338 338 338 338 338 338 338 338 339 339 339 339 339 339 339 339 339 339
## [4771] 339 339 339 339 339 339 339 339 339 339 340 340 340 340 340 340 340 340
## [4789] 340 340 340 340 340 340 340 340 340 340 340 340 341 341 341 341 341 341
## [4807] 341 341 341 341 341 341 341 341 341 341 341 341 341 341 342 342 342 342
## [4825] 342 342 342 342 342 342 342 342 342 342 342 342 342 342 342 342 343 343
## [4843] 343 343 343 343 343 343 343 343 343 343 343 343 343 343 343 343 343 343
## [4861] 344 344 344 344 344 344 344 344 344 344 344 344 344 344 344 344 344 344
## [4879] 344 344 345 345 345 345 345 345 345 345 345 345 345 345 345 345 345 345
## [4897] 345 345 345 345 346 346 346 346 346 346 346 346 346 346 346 346 346 346
## [4915] 346 346 346 346 346 346 347 347 347 347 347 347 347 347 347 347 347 347
## [4933] 347 347 347 347 347 347 347 347 348 348 348 348 348 348 348 348 348 348
## [4951] 348 348 348 348 348 348 348 348 348 348 349 349 349 349 349 349 349 349
## [4969] 349 349 349 349 349 349 349 349 349 349 349 349 350 350 350 350 350 350
## [4987] 350 350 350 350 350 350 350 350 350 350 350 350 350 350 351 351 351 351
## [5005] 351 351 351 351 351 351 351 351 351 351 351 351 351 351 351 351 352 352
## [5023] 352 352 352 352 352 352 352 352 352 352 352 352 352 352 352 352 352 352
## [5041] 353 353 353 353 353 353 353 353 353 353 353 353 353 353 353 353 353 353
## [5059] 353 353 354 354 354 354 354 354 354 354 354 354 354 354 354 354 354 354
## [5077] 354 354 354 354 355 355 355 355 355 355 355 355 355 355 355 355 355 355
## [5095] 355 355 355 355 355 355 356 356 356 356 356 356 356 356 356 356 356 356
## [5113] 356 356 356 356 356 356 356 356 357 357 357 357 357 357 357 357 357 357
## [5131] 357 357 357 357 357 357 357 357 357 357 358 358 358 358 358 358 358 358
## [5149] 358 358 358 358 358 358 358 358 358 358 358 358 359 359 359 359 359 359
## [5167] 359 359 359 359 359 359 359 359 359 359 359 359 359 359 360 360 360 360
## [5185] 360 360 360 360 360 360 360 360 360 360 360 360 360 360 360 360 361 361
## [5203] 361 361 361 361 361 361 361 361 361 361 361 361 361 361 361 361 361 361
## [5221] 362 362 362 362 362 362 362 362 362 362 362 362 362 362 362 362 362 362
## [5239] 362 362 363 363 363 363 363 363 363 363 363 363 363 363 363 363 363 363
## [5257] 363 363 363 363 364 364 364 364 364 364 364 364 364 364 364 364 364 364
## [5275] 364 364 364 364 364 364 365 365 365 365 365 365 365 365 365 365 365 365
## [5293] 365 365 365 365 365 365 365 365 366 366 366 366 366 366 366 366 366 366
## [5311] 366 366 366 366 366 366 366 366 366 366 367 367 367 367 367 367 367 367
## [5329] 367 367 367 367 367 367 367 367 367 367 367 367 368 368 368 368 368 368
## [5347] 368 368 368 368 368 368 368 368 368 368 368 368 368 368 369 369 369 369
## [5365] 369 369 369 369 369 369 369 369 369 369 369 369 369 369 369 369 370 370
## [5383] 370 370 370 370 370 370 370 370 370 370 370 370 370 370 370 370 370 370
## [5401] 371 371 371 371 371 371 371 371 371 371 371 371 371 371 371 371 371 371
## [5419] 371 371 372 372 372 372 372 372 372 372 372 372 372 372 372 372 372 372
## [5437] 372 372 372 372 373 373 373 373 373 373 373 373 373 373 373 373 373 373
## [5455] 373 373 373 373 373 373 374 374 374 374 374 374 374 374 374 374 374 374
## [5473] 374 374 374 374 374 374 374 374 375 375 375 375 375 375 375 375 375 375
## [5491] 375 375 375 375 375 375 375 375 375 375 376 376 376 376 376 376 376 376
## [5509] 376 376 376 376 376 376 376 376 376 376 376 376 377 377 377 377 377 377
## [5527] 377 377 377 377 377 377 377 377 377 377 377 377 377 377 378 378 378 378
## [5545] 378 378 378 378 378 378 378 378 378 378 378 378 378 378 378 378 379 379
## [5563] 379 379 379 379 379 379 379 379 379 379 379 379 379 379 379 379 379 379
## [5581] 380 380 380 380 380 380 380 380 380 380 380 380 380 380 380 380 380 380
## [5599] 380 380 381 381 381 381 381 381 381 381 381 381 381 381 381 381 381 381
## [5617] 381 381 381 381 382 382 382 382 382 382 382 382 382 382 382 382 382 382
## [5635] 382 382 382 382 382 382 383 383 383 383 383 383 383 383 383 383 383 383
## [5653] 383 383 383 383 383 383 383 383 384 384 384 384 384 384 384 384 384 384
## [5671] 384 384 384 384 384 384 384 384 384 384 385 385 385 385 385 385 385 385
## [5689] 385 385 385 385 385 385 385 385 385 385 385 385 386 386 386 386 386 386
## [5707] 386 386 386 386 386 386 386 386 386 386 386 386 386 386 387 387 387 387
## [5725] 387 387 387 387 387 387 387 387 387 387 387 387 387 387 387 387 388 388
## [5743] 388 388 388 388 388 388 388 388 388 388 388 388 388 388 388 388 388 388
## [5761] 389 389 389 389 389 389 389 389 389 389 389 389 389 389 389 389 389 389
## [5779] 389 389 390 390 390 390 390 390 390 390 390 390 390 390 390 390 390 390
## [5797] 390 390 390 390 391 391 391 391 391 391 391 391 391 391 391 391 391 391
## [5815] 391 391 391 391 391 391 392 392 392 392 392 392 392 392 392 392 392 392
## [5833] 392 392 392 392 392 392 392 392 393 393 393 393 393 393 393 393 393 393
## [5851] 393 393 393 393 393 393 393 393 393 393 394 394 394 394 394 394 394 394
## [5869] 394 394 394 394 394 394 394 394 394 394 394 394 395 395 395 395 395 395
## [5887] 395 395 395 395 395 395 395 395 395 395 395 395 395 395 396 396 396 396
## [5905] 396 396 396 396 396 396 396 396 396 396 396 396 396 396 396 396 397 397
## [5923] 397 397 397 397 397 397 397 397 397 397 397 397 397 397 397 397 397 397
## [5941] 398 398 398 398 398 398 398 398 398 398 398 398 398 398 398 398 398 398
## [5959] 398 398 399 399 399 399 399 399 399 399 399 399 399 399 399 399 399 399
## [5977] 399 399 399 399 400 400 400 400 400 400 400 400 400 400 400 400 400 400
## [5995] 400 400 400 400 400 400А если весь датасет?
7.8.2 arrange()
Тогда вот так:
## # A tibble: 6,000 x 10
## trialtype setsize numtrial time1 click1x click1y time2 click2x click2y id
## <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <int>
## 1 tray 8 1 1.67 -227 202 1.28 14 -351 1
## 2 tray 8 1 2.25 -252 198 2.53 28 -362 2
## 3 tray 8 1 1.04 -212 211 0.969 27 -384 3
## 4 tray 8 1 1.32 -244 199 0.666 -15 -408 4
## 5 tray 8 1 0.905 -255 190 0.361 150 -369 5
## 6 tray 8 1 2.45 6 -357 2.27 83 36 6
## 7 tray 8 1 1.44 -224 175 0.797 31 -345 7
## 8 tray 8 1 1.49 -243 219 1.05 2 -378 8
## 9 tray 8 1 1.10 -232 198 0.781 42 -403 9
## 10 tray 8 1 0.981 -226 202 0.582 -21 -372 10
## # … with 5,990 more rowsИли по двум переменным сразу:
## # A tibble: 6,000 x 10
## trialtype setsize numtrial time1 click1x click1y time2 click2x click2y id
## <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <int>
## 1 tray 8 1 1.67 -227 202 1.28 14 -351 1
## 2 tray 8 1 2.25 -252 198 2.53 28 -362 2
## 3 tray 8 1 1.04 -212 211 0.969 27 -384 3
## 4 tray 8 1 1.32 -244 199 0.666 -15 -408 4
## 5 tray 8 1 0.905 -255 190 0.361 150 -369 5
## 6 tray 8 1 2.45 6 -357 2.27 83 36 6
## 7 tray 8 1 1.44 -224 175 0.797 31 -345 7
## 8 tray 8 1 1.49 -243 219 1.05 2 -378 8
## 9 tray 8 1 1.10 -232 198 0.781 42 -403 9
## 10 tray 8 1 0.981 -226 202 0.582 -21 -372 10
## # … with 5,990 more rowsВ данном случае получилось то же самое, так как мы присоединяли данные испытуемых последовательно с увеличивая id.
7.8.3 distinct()
Можно вывести уникальные сочетания по двум переменным:
## # A tibble: 6 x 2
## trialtype setsize
## <chr> <dbl>
## 1 tray 8
## 2 dots 8
## 3 tray 12
## 4 dots 12
## 5 tray 16
## 6 dots 16Тут все достаточно ожидаемо — (квази)эксперимент всё-таки.
Так, погодите! А если это квазиэксперимент, то где же квазиНП? Она точно была! В описании эксперимента фигурировала используемая платформа смартфона!
Спокойно. Ща всё будет.
7.9 Соединение датафреймов II
Данные о том, смартфон на какой платформе использует испытуемый, были записаны в отдельный датасет. Он вот тут.
Грузим его:
platform <- read_csv('https://raw.githubusercontent.com/angelgardt/hseuxlab-wlm2021/master/book/wlm2021-book/data/share_platform.csv')##
## ── Column specification ────────────────────────────────────────────────────────
## cols(
## id = col_double(),
## platform = col_character()
## )Осталось понять, как нам его присобачить к имеющимся данным так, как нам надо. А нам надо так, чтобы появилась новая колонка, в каждой ячейке которой согласно id будет стоять соответствующая платформа.
7.9.1 ..._join()
Нас спасёт ..._join(). Эта функция объединяет два датасета по определенной ключевой переменной.
Что делает ..._join()? У него есть две ключевые колонки из двух датасетов. Он находит соответствия, и лепит все колонки одной таблицы к другой, сопоставляя соответствующие строки.
- Для
left_join()главный тиббл — слева, то есть остаются все строки левого тиббла, а из правого выбираются только те, которые нужны. - Для
right_join()главный тиббл — справа, то есть остаются все строки правого тиббла, а из левого выбираются те, которые нужны. - Для
full_join()иinner_join()главные тибблы оба:full_join()оставляет все строки обоих тибблов, а те, которым не нашлось соответствия в другом тиббле, заполняетNA.inner_join()оставляет только те строки, которым нашлось соответствие, а всё остальные удаляет.
anti_join()оставляет только те строки, которым не нашлось соответствия.
## Joining, by = "id"## # A tibble: 5 x 3
## id x1 x2
## <dbl> <chr> <chr>
## 1 1 A <NA>
## 2 2 B K
## 3 3 C <NA>
## 4 4 D L
## 5 5 E <NA>## Joining, by = "id"## # A tibble: 5 x 3
## id x1 x2
## <dbl> <chr> <chr>
## 1 2 B K
## 2 4 D L
## 3 6 <NA> M
## 4 8 <NA> N
## 5 10 <NA> O## Joining, by = "id"## # A tibble: 8 x 3
## id x1 x2
## <dbl> <chr> <chr>
## 1 1 A <NA>
## 2 2 B K
## 3 3 C <NA>
## 4 4 D L
## 5 5 E <NA>
## 6 6 <NA> M
## 7 8 <NA> N
## 8 10 <NA> O## Joining, by = "id"## # A tibble: 2 x 3
## id x1 x2
## <dbl> <chr> <chr>
## 1 2 B K
## 2 4 D L## Joining, by = "id"## # A tibble: 3 x 2
## id x1
## <int> <chr>
## 1 1 A
## 2 3 C
## 3 5 EКак нам это поможет? У нас есть колонка id в обоих датасетах — share и platform. По ней мы и сможем соединить наши тибблы. Нам надо сделать так, чтобы все наши экспериментальные данные были сохранны. Используем left_join():
## Joining, by = "id"## # A tibble: 6,000 x 11
## trialtype setsize numtrial time1 click1x click1y time2 click2x click2y id
## <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 tray 8 1 1.67 -227 202 1.28 14 -351 1
## 2 tray 8 2 1.13 -69 231 1.06 -44 -392 1
## 3 tray 8 3 2.60 60 195 0.963 17 -361 1
## 4 tray 8 4 2.61 199 213 0.863 -26 -356 1
## 5 tray 8 5 1.59 -241 43 0.931 -25 -397 1
## 6 tray 8 6 1.14 -51 59 0.896 10 -383 1
## 7 tray 8 7 2.14 99 62 0.879 -29 -372 1
## 8 tray 8 8 1.63 213 46 0.947 -27 -353 1
## 9 tray 8 9 2.56 -201 -123 1.65 -25 -385 1
## 10 tray 8 10 2.02 -70 -82 0.970 -19 -394 1
## # … with 5,990 more rows, and 1 more variable: platform <chr>Наблюдаем, что строки platform были продублированы столько раз, сколько нужно было, чтобы сохранились все строки тиббла с экспериментальными данными.
Не забываем перезаписать наш тиббл:
## Joining, by = "id"
В нашей папке с данными есть файл targetpositions.xlsx, который содержит координаты, по которым мы сможем отобрать правильные клики наших испытуемых. Изучите этот файл, загрузите его как tibble и соедините его с нашим основным тибблом share.
Подумайте, какие колонки тибблов вы будете использовать для объединения.
## # A tibble: 6,150 x 23
## trialtype setsize numtrial time1 click1x click1y time2 click2x click2y id
## <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 tray 8 1 1.67 -227 202 1.28 14 -351 1
## 2 tray 8 2 1.13 -69 231 1.06 -44 -392 1
## 3 tray 8 3 2.60 60 195 0.963 17 -361 1
## 4 tray 8 4 2.61 199 213 0.863 -26 -356 1
## 5 tray 8 5 1.59 -241 43 0.931 -25 -397 1
## 6 tray 8 6 1.14 -51 59 0.896 10 -383 1
## 7 tray 8 7 2.14 99 62 0.879 -29 -372 1
## 8 tray 8 8 1.63 213 46 0.947 -27 -353 1
## 9 tray 8 9 2.56 -201 -123 1.65 -25 -385 1
## 10 tray 8 10 2.02 -70 -82 0.970 -19 -394 1
## # … with 6,140 more rows, and 13 more variables: platform <chr>, posx1 <dbl>,
## # posy1 <dbl>, posxmin1 <dbl>, posxmax1 <dbl>, posymin1 <dbl>,
## # posymax1 <dbl>, posx2 <dbl>, posy2 <dbl>, posxmin2 <dbl>, posxmax2 <dbl>,
## # posymin2 <dbl>, posymax2 <dbl>7.10 Группировка и аггрегация данных
Часто возникает необходимость аггрегировать данные, то есть посчитать какие-то показатели, причем на по всему датасету, а по группам, имеющимся в нём. Займемся этим мероприятием.
7.10.1 group_by() & ungroup()
Нам тиббл сам по себе не знает, что в нём есть какие-то группы. Поэтому ему надо рассказать, как дело обстоит. Сгруппируем наши данные по экспериметальным условиям:
## # A tibble: 6,150 x 23
## # Groups: trialtype [3]
## trialtype setsize numtrial time1 click1x click1y time2 click2x click2y id
## <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 tray 8 1 1.67 -227 202 1.28 14 -351 1
## 2 tray 8 2 1.13 -69 231 1.06 -44 -392 1
## 3 tray 8 3 2.60 60 195 0.963 17 -361 1
## 4 tray 8 4 2.61 199 213 0.863 -26 -356 1
## 5 tray 8 5 1.59 -241 43 0.931 -25 -397 1
## 6 tray 8 6 1.14 -51 59 0.896 10 -383 1
## 7 tray 8 7 2.14 99 62 0.879 -29 -372 1
## 8 tray 8 8 1.63 213 46 0.947 -27 -353 1
## 9 tray 8 9 2.56 -201 -123 1.65 -25 -385 1
## 10 tray 8 10 2.02 -70 -82 0.970 -19 -394 1
## # … with 6,140 more rows, and 13 more variables: platform <chr>, posx1 <dbl>,
## # posy1 <dbl>, posxmin1 <dbl>, posxmax1 <dbl>, posymin1 <dbl>,
## # posymax1 <dbl>, posx2 <dbl>, posy2 <dbl>, posxmin2 <dbl>, posxmax2 <dbl>,
## # posymin2 <dbl>, posymax2 <dbl>Теперь в выводе тиббла указаны переменные, по которым сгруппированы наши данные (Groups). В структуре это тоже будет отображено:
## tibble [6,150 × 23] (S3: grouped_df/tbl_df/tbl/data.frame)
## $ trialtype: chr [1:6150] "tray" "tray" "tray" "tray" ...
## $ setsize : num [1:6150] 8 8 8 8 8 8 8 8 8 8 ...
## $ numtrial : num [1:6150] 1 2 3 4 5 6 7 8 9 10 ...
## $ time1 : num [1:6150] 1.67 1.13 2.6 2.61 1.59 ...
## $ click1x : num [1:6150] -227 -69 60 199 -241 -51 99 213 -201 -70 ...
## $ click1y : num [1:6150] 202 231 195 213 43 59 62 46 -123 -82 ...
## $ time2 : num [1:6150] 1.28 1.061 0.963 0.863 0.931 ...
## $ click2x : num [1:6150] 14 -44 17 -26 -25 10 -29 -27 -25 -19 ...
## $ click2y : num [1:6150] -351 -392 -361 -356 -397 -383 -372 -353 -385 -394 ...
## $ id : num [1:6150] 1 1 1 1 1 1 1 1 1 1 ...
## $ platform : chr [1:6150] "ios" "ios" "ios" "ios" ...
## $ posx1 : num [1:6150] -238 -63 66 203 -243 -60 73 213 -229 -84 ...
## $ posy1 : num [1:6150] 202 226 217 218 59 90 66 52 -93 -79 ...
## $ posxmin1 : num [1:6150] -313 -138 -9 128 -318 -135 -2 138 -304 -159 ...
## $ posxmax1 : num [1:6150] -163 12 141 278 -168 15 148 288 -154 -9 ...
## $ posymin1 : num [1:6150] 127 151 142 143 -16 15 -9 -23 -168 -154 ...
## $ posymax1 : num [1:6150] 277 301 292 293 134 165 141 127 -18 -4 ...
## $ posx2 : num [1:6150] 450 450 450 450 450 450 450 450 450 450 ...
## $ posy2 : num [1:6150] -350 -350 -350 -350 -350 -350 -350 -350 -350 -350 ...
## $ posxmin2 : num [1:6150] 350 350 350 350 350 350 350 350 350 350 ...
## $ posxmax2 : num [1:6150] 550 550 550 550 550 550 550 550 550 550 ...
## $ posymin2 : num [1:6150] -425 -425 -425 -425 -425 -425 -425 -425 -425 -425 ...
## $ posymax2 : num [1:6150] -275 -275 -275 -275 -275 -275 -275 -275 -275 -275 ...
## - attr(*, "groups")= tibble [3 × 2] (S3: tbl_df/tbl/data.frame)
## ..$ trialtype: chr [1:3] "dots" "tray" NA
## ..$ .rows : list<int> [1:3]
## .. ..$ : int [1:3000] 51 52 53 54 55 56 57 58 59 60 ...
## .. ..$ : int [1:3000] 1 2 3 4 5 6 7 8 9 10 ...
## .. ..$ : int [1:150] 6001 6002 6003 6004 6005 6006 6007 6008 6009 6010 ...
## .. ..@ ptype: int(0)
## ..- attr(*, ".drop")= logi TRUEВ первой строчке аутпута появилось указание groupped. Это как раз то, что мы хотели сделать. Можно перезаписать наш тиббл и посмотреть, как он будет выводиться в консоль:
Если нам нужно убрать группировку, то есть функция ungroup():
## # A tibble: 6,150 x 23
## trialtype setsize numtrial time1 click1x click1y time2 click2x click2y id
## <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 tray 8 1 1.67 -227 202 1.28 14 -351 1
## 2 tray 8 2 1.13 -69 231 1.06 -44 -392 1
## 3 tray 8 3 2.60 60 195 0.963 17 -361 1
## 4 tray 8 4 2.61 199 213 0.863 -26 -356 1
## 5 tray 8 5 1.59 -241 43 0.931 -25 -397 1
## 6 tray 8 6 1.14 -51 59 0.896 10 -383 1
## 7 tray 8 7 2.14 99 62 0.879 -29 -372 1
## 8 tray 8 8 1.63 213 46 0.947 -27 -353 1
## 9 tray 8 9 2.56 -201 -123 1.65 -25 -385 1
## 10 tray 8 10 2.02 -70 -82 0.970 -19 -394 1
## # … with 6,140 more rows, and 13 more variables: platform <chr>, posx1 <dbl>,
## # posy1 <dbl>, posxmin1 <dbl>, posxmax1 <dbl>, posymin1 <dbl>,
## # posymax1 <dbl>, posx2 <dbl>, posy2 <dbl>, posxmin2 <dbl>, posxmax2 <dbl>,
## # posymin2 <dbl>, posymax2 <dbl>7.10.2 summarize()
Сама по себе функция group_by() особого смысла не имеет, но вот есть её использовать в связке в summarize()…
## `summarise()` ungrouping output (override with `.groups` argument)## # A tibble: 3 x 3
## trialtype mean sd
## <chr> <dbl> <dbl>
## 1 dots 1.60 0.886
## 2 tray 1.65 0.896
## 3 <NA> NA NAЧто мы здесь сделали? Мы хотели посчитать среднее и стандартное отклонение времени реакции (первого клика), но не по всему датасету, а по экспериментальным условиям. Для этого мы сгруппироали тиббл по переменной, содержащей указания на экспериментальное условие, и затем аггрегировали наши данные — посчитали по ним некоторые статистики. Функция §ummarise() возвращает тиббл, в котором по строкам идут заданные нами группы, а по столбцам те статистики, которые мы указали в её аргументах. Для их подсчёта можно использовать любые функции — как специализированные их пакетов, так и встроенные в базовый R.
Допустим, мы хотим проверить, что эксперимент был сбалансирован по количеству проб в разных условиях. Тогда сделаем так:
## `summarise()` regrouping output by 'trialtype' (override with `.groups` argument)## # A tibble: 7 x 3
## # Groups: trialtype [3]
## trialtype setsize n
## <chr> <dbl> <int>
## 1 dots 8 1000
## 2 dots 12 1000
## 3 dots 16 1000
## 4 tray 8 1000
## 5 tray 12 1000
## 6 tray 16 1000
## 7 <NA> NA 150
При предобработке данных нам часто необходимо усреднить экспериментальные пробы по респондентам. Возьмите имеющиеся данные share и подготовьте их к дисперсионному анализу.
Создайте новую логическую переменную, которая будет содержать информацию о том, корректно ли выполнена данная проба. Корректной считается проба, в которой координаты клика респондента (
click1x,click1y) попали в целевые диапазоны ([posxmin1,posxmax1] и [posymin1,posymax1]).Отберите только корректные пробы.
Рассчитайте среднее и стандартное отклонение времени реакции (
time1) по экспериментальным условиям (trialtype,setsize) для каждого испытуемого. Итоговый тиббл также должен содержать колонкуplatform.Сохраните результат в новый объект.
## `summarise()` regrouping output by 'id', 'trialtype', 'setsize' (override with `.groups` argument)## # A tibble: 120 x 6
## # Groups: id, trialtype, setsize [120]
## id trialtype setsize platform mean sd
## <dbl> <chr> <dbl> <chr> <dbl> <dbl>
## 1 1 dots 8 ios 1.59 0.467
## 2 1 dots 12 ios 1.88 0.647
## 3 1 dots 16 ios 2.18 1.14
## 4 1 tray 8 ios 1.56 0.453
## 5 1 tray 12 ios 1.77 0.739
## 6 1 tray 16 ios 2.12 1.08
## 7 2 dots 8 ios 1.54 0.566
## 8 2 dots 12 ios 1.59 0.613
## 9 2 dots 16 ios 1.99 1.00
## 10 2 tray 8 ios 1.44 0.446
## # … with 110 more rows7.11 Широкий и длинный формат
Мы получили новый тиббл который содержит аггрегированные данные. Теперь нам надо поговорить о широком и длинном форматах данных.
Как мы говорили ранее, tidy data задаёт нам следующий формат данных: по строкам идут наблюдения, по столбцам — отдельные переменные. Такой формат данных называется широким. Широкий он потому, что у нас может быть много отдельных переменных. Длиннный формат данных выглядит следующим образом: есть столбец индентификаторов (например, ID испытуемого), есть столбец переменных, и есть столбец значений этих переменных.
Это общая логика, и под каждую задачу у нас может быть свой длинный и свой широкий формат. Кроме того, данные могут быть разной «широкости» и разной «длинности», что тоже определяется конкретной задачей.
7.11.1 pivot_longer() & pivot_wider()
Наши данные сейчас — «средние». С одно стороны он — широкий, так как у нас есть две переменные mean и id, которые записаны в отдельных столбцах:
## # A tibble: 120 x 6
## # Groups: id, trialtype, setsize [120]
## id trialtype setsize platform mean sd
## <dbl> <chr> <dbl> <chr> <dbl> <dbl>
## 1 1 dots 8 ios 1.59 0.467
## 2 1 dots 12 ios 1.88 0.647
## 3 1 dots 16 ios 2.18 1.14
## 4 1 tray 8 ios 1.56 0.453
## 5 1 tray 12 ios 1.77 0.739
## 6 1 tray 16 ios 2.12 1.08
## 7 2 dots 8 ios 1.54 0.566
## 8 2 dots 12 ios 1.59 0.613
## 9 2 dots 16 ios 1.99 1.00
## 10 2 tray 8 ios 1.44 0.446
## # … with 110 more rowsМы можем перевести наши данный в длинный формат:
## # A tibble: 240 x 6
## # Groups: id, trialtype, setsize [120]
## id trialtype setsize platform name value
## <dbl> <chr> <dbl> <chr> <chr> <dbl>
## 1 1 dots 8 ios mean 1.59
## 2 1 dots 8 ios sd 0.467
## 3 1 dots 12 ios mean 1.88
## 4 1 dots 12 ios sd 0.647
## 5 1 dots 16 ios mean 2.18
## 6 1 dots 16 ios sd 1.14
## 7 1 tray 8 ios mean 1.56
## 8 1 tray 8 ios sd 0.453
## 9 1 tray 12 ios mean 1.77
## 10 1 tray 12 ios sd 0.739
## # … with 230 more rowsУ нас появилась колонка name, которая задаёт названия наших переменных, и колонка value, которая задаёт их значения.
Однако мы можем перевести исходный датасет и в более широкий формат:
share_aggregated %>% select(-sd) %>% # переменная sd будет лишней
pivot_wider(names_from = 'setsize', values_from = 'mean')## # A tibble: 40 x 6
## # Groups: id, trialtype [40]
## id trialtype platform `8` `12` `16`
## <dbl> <chr> <chr> <dbl> <dbl> <dbl>
## 1 1 dots ios 1.59 1.88 2.18
## 2 1 tray ios 1.56 1.77 2.12
## 3 2 dots ios 1.54 1.59 1.99
## 4 2 tray ios 1.44 1.74 2.68
## 5 3 dots ios 1.53 1.82 1.99
## 6 3 tray ios 1.46 2.05 2.48
## 7 4 dots andr 1.35 1.73 1.97
## 8 4 tray andr 1.32 1.61 1.65
## 9 5 dots ios 1.22 1.34 1.44
## 10 5 tray ios 1.22 1.51 1.73
## # … with 30 more rowsНаблюдаем, что теперь время в различных сетсайзах расположено в отдельных колонках.
Из широкого формата удобнее доставать отдельные переменные, например, для сравнения t-тестов. Из длинного формата удобно доставать вектор значений и вектор групп, например, для дисперсионного анализа. Мы будем работать и с тем и с другим форматом, поэтому перемещаться между ними надо быстро и уверенно.
Помните разговор о пространстве имен?↩︎
Чтобы полностью понять вторую строку, надо вспомнить, что оператор деления
/— это тоже функция, которая принимает в себя два аргумента. В данном случае нам надо поделить единицу на косинус, значит косинус должен идти вторым.↩︎